Identificación de genes codificantes y elementos repetidos regulados por DAXX mediante RNA-SEQ
 Effects of DAXX downregulation
Effects of DAXX downregulation
Abstract
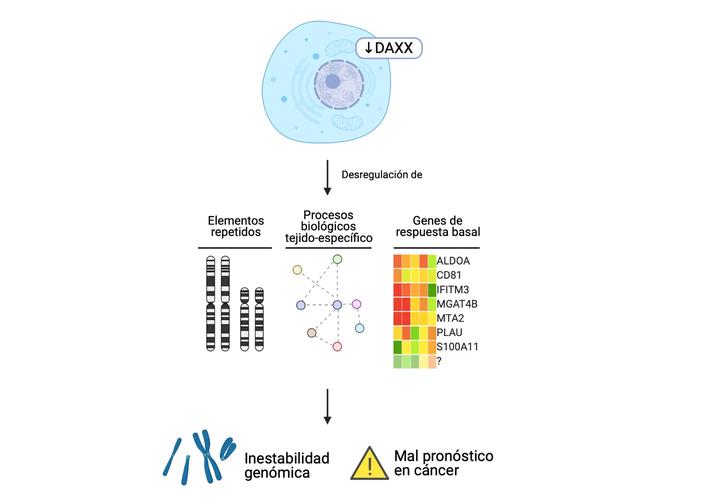
La célula eucarionte posee mecanismos de regulación epigenética que le permiten modular los genes que se expresan en un momento dado. Con ayuda de estos mecanismos, esta es capaz de responder a los estímulos y generar un perfil de expresión que se adapte al ambiente y preserve su identidad celular. Dentro de las capas de regulación pre-transcripcional, las variantes de histonas juegan un papel muy importante al formar nucleosomas especializados. Estas estructuras desempeñan funciones específicas en el núcleo celular, tales como mantenimiento de la heterocromatina, apertura transcripcional, formación del cromosoma mitótico, entre otras. Uno de los elementos más importantes para que las variantes de histonas cumplan su función de manera correcta son las chaperonas. Estas macromoléculas se encargan de depositar al nucleosoma variante en las regiones correctas del genoma. A su vez, estas pueden asociarse con factores epigenéticos que modifican el epigenoma circundante y desencadenan la función celular asociada con la presencia de dicho nucleosoma especializado. En este contexto, la chaperona DAXX se encarga de depositar a la variante de histona H3.3 en el genoma. Esto se ha visto asociado principalmente con represión transcripcional de elementos repetitivos y genes codificantes, así como en el mantenimiento de los dominios de heterocromatina en el núcleo celular. Además, la alteración en la expresión de DAXX se ha asociado con inestabilidad cromosómica y fenotipos agresivos in vivo e in vitro en distintos tipos de cáncer. A pesar de esto, pocos estudios han descrito los genes codificantes regulados por esta chaperona, y ninguno ha explorado los elementos repetitivos involucrados en este fenómeno. En esta tesis, se analizaron datos de secuenciación de RNA en tres líneas celulares con el objetivo de describir los principales elementos alterados tras la represión de esta proteína. Al analizar los procesos biológicos desregulados, se encontró que DAXX modula principalmente genes tejido específico en cada línea celular. Aún así, se encontraron genes sobre-expresados en común que componen una firma basal en todos los tejidos estudiados. Todos ellos son importantes en el desarrollo del cáncer, como ALDOA, PLAU, MGAT4B y S100A11. Este patrón se repitió en los elementos repetitivos, donde la intersección entre cada experimento fue pequeña o nula; aún así, cada línea celular presentó repetidos desregulados como ACRO1 en HCT116, TAR1 en PC3 o MSTB2 en GSC23. Sorprendentemente, encontramos datos que sugieren que DAXX está involucrado también en la activación transcripcional. Los resultados sugieren un mecanismo en el cual DAXX se asocia a factores transcripcionales tejido específico para modular genes en la célula; no obstante, algunos pocos son regulados independientemente del tipo celular. Estos resultados son particularmente relevantes para identificar elementos importantes en el fenotipo agresivo ocasionado por la desregulación de DAXX en cáncer.